Général
Un nouveau modèle d’IA révolutionnaire prédit avec précision l’interaction entre protéines et ADN !
Une avancée révolutionnaire dans le domaine de la biologie moléculaire a été réalisée avec le modèle d’intelligence artificielle Deep Predictor of Binding Specificity (DeepPBS), développé par des chercheurs de l’USC. Ce modèle de deep learning géométrique prédit avec précision comment une variété de protéines se lient à l’ADN, ouvrant la voie à de nouvelles thérapies médicales. Grâce à DeepPBS, les scientifiques peuvent désormais analyser les interactions protéine-ADN de manière intuitive, transformant ainsi notre compréhension de la régulation génétique et accélérant le développement de médicaments ciblés.

Une avancée majeure dans la prédiction de la spécificité de liaison des protéines
Des chercheurs de l’Université de Californie du Sud (USC) ont mis au point un modèle d’intelligence artificielle révolutionnaire, baptisé Deep Predictor of Binding Specificity (DeepPBS), qui a été publié dans la prestigieuse revue Nature Methods. Ce modèle de pointe permet de prédire avec précision la manière dont une large gamme de protéines interagit avec l’ADN, marquant ainsi une avancée significative dans le développement de nouveaux médicaments et traitements médicaux.
Un modèle innovant pour l’analyse des interactions protéine-ADN
DeepPBS est un modèle de deep learning géométrique conçu pour anticiper la spécificité de liaison des protéines à l’ADN en se basant sur des structures complexes. Cet outil puissant offre aux scientifiques la possibilité d’entrer les données structurelles d’un complexe protéine-ADN dans une plateforme de calcul en ligne intuitive, transformant ainsi le domaine de l’analyse des interactions protéine-ADN.
Importance de la spécificité de liaison dans la régulation génétique
« Les structures des complexes protéine-ADN contiennent généralement des protéines liées à une séquence d’ADN unique. Pour comprendre la régulation des gènes, il est essentiel d’accéder à la spécificité de liaison d’une protéine à n’importe quelle séquence ou région du génome, » a déclaré Remo Rohs, professeur et président fondateur du Département de biologie quantitative et computationnelle à l’USC Dornsife College of Letters, Arts and Sciences.
« DeepPBS est un outil d’IA qui élimine le besoin de séquençage à haut débit ou d’expériences de biologie structurale pour révéler la spécificité de liaison protéine-ADN. »
Une technologie de pointe pour des prédictions précises
DeepPBS exploite un modèle de deep learning géométrique, une forme d’apprentissage automatique qui analyse les données à l’aide de structures géométriques. Cet outil d’IA avancé est spécifiquement conçu pour examiner les propriétés chimiques et les contextes géométriques des interactions protéine-ADN afin de prédire avec précision la spécificité de liaison.
En analysant ces données, DeepPBS génère des graphes spatiaux qui représentent visuellement la structure des protéines et l’interaction complexe entre les protéines et l’ADN. Contrairement à de nombreuses méthodes existantes, souvent limitées à des familles de protéines spécifiques, DeepPBS peut prédire la spécificité de liaison à travers diverses familles de protéines, ce qui en fait un outil polyvalent et puissant en bioinformatique.
Un outil universel pour la recherche
« Il est crucial que les chercheurs disposent d’une méthode applicable à toutes les protéines, sans se limiter à une famille de protéines bien étudiée. Cette approche nous permet également de concevoir de nouvelles protéines, » a ajouté Rohs.
Des avancées dans la prédiction de la structure des protéines
Le domaine de la prédiction de la structure des protéines a connu des progrès significatifs grâce à AlphaFold de DeepMind, qui peut prédire la structure des protéines à partir de données de séquence. Ces avancées ont permis d’accumuler une grande quantité de données structurelles accessibles pour l’analyse scientifique. En combinant DeepPBS avec des méthodes de prédiction de structure, il est possible de prédire la spécificité des protéines sans structures expérimentales.
Applications prometteuses de DeepPBS
Rohs a souligné les nombreuses applications potentielles de DeepPBS. Cet outil de recherche révolutionnaire pourrait accélérer le développement de nouveaux médicaments et traitements adaptés à des mutations spécifiques dans les cellules cancéreuses. De plus, il pourrait ouvrir la voie à de nouvelles découvertes en biologie synthétique et trouver des applications dans la recherche sur l’ARN.
Référence de l’article
- Raktim Mitra, Jinsen Li, Jared M. Sagendorf, Yibei Jiang, Ari S. Cohen, Tsu-Pei Chiu, Cameron J. Glasscock & Remo Rohs. Apprentissage profond géométrique de la spécificité de liaison protéine-ADN. Nature Methods, 2024; DOI: 10.1038/s41592-024-02372-w
Général
Anker SOLIX dévoile la Solarbank 2 AC : la nouvelle ère du stockage d’énergie ultra-compatible !
Découvrez le Solarbank 2 AC, une véritable révolution dans le domaine de l’énergie solaire ! Grâce à ses batteries au phosphate de fer lithium, ce système s’adapte parfaitement à vos besoins. Avec une puissance impressionnante de 2400 watts et la possibilité d’ajouter jusqu’à cinq batteries supplémentaires, il assure un stockage optimal. Sa compatibilité avec le compteur Anker SOLIX Smart favorise une gestion intelligente de votre consommation énergétique. Ne ratez pas l’offre spéciale « early bird », disponible dès maintenant pour seulement 999 euros ! Saisissez cette chance unique !

Le Solarbank 2 AC : Une Révolution dans le Stockage Énergétique
Batteries au Lithium Fer Phosphate
Le Solarbank 2 AC se démarque par l’utilisation de batteries au lithium fer phosphate (LFP), reconnues pour leur sécurité et leur efficacité. Ce modèle est particulièrement innovant grâce à son système de couplage alternatif, qui lui permet de s’adapter facilement à divers systèmes solaires déjà en place.Que ce soit pour des installations sur toiture, des systèmes solaires compacts pour balcons ou d’autres configurations réduites, il peut fonctionner avec un micro-onduleur de 800 Watts.
Capacité et flexibilité Énergétique
Avec une capacité maximale d’injection dans le réseau domestique atteignant 1200 watts,le Solarbank 2 AC peut être associé à deux régulateurs solaires MPPT. Cela ouvre la possibilité d’ajouter jusqu’à 1200 watts supplémentaires via des panneaux solaires additionnels, portant ainsi la puissance totale à un impressionnant 2400 watts. Pour les utilisateurs nécessitant davantage de stockage énergétique, il est possible d’intégrer jusqu’à cinq batteries supplémentaires de 1,6 kilowattheure chacune, augmentant la capacité totale à 9,6 kilowattheures.
Intégration dans un Écosystème Intelligent
Le Solarbank 2 AC s’intègre parfaitement dans un écosystème énergétique intelligent grâce à sa compatibilité avec le compteur Anker SOLIX Smart et les prises intelligentes proposées par Anker. cette fonctionnalité permet une gestion optimisée de la consommation électrique tout en réduisant les pertes énergétiques inutiles. De plus, Anker SOLIX prévoit d’étendre cette compatibilité aux dispositifs Shelly.
Durabilité et Résistance aux Intempéries
Anker SOLIX met également l’accent sur la longévité du Solarbank 2 AC. Conçu pour supporter au moins 6000 cycles de charge, cet appareil a une durée de vie estimée dépassant quinze ans. Il est accompagné d’une garantie fabricant décennale et possède une certification IP65 qui assure sa résistance face aux intempéries tout en étant capable de fonctionner dans des températures variant entre -20 °C et +55 °C.
Disponibilité et Offres Promotionnelles
Le solarbank 2 AC est disponible sur le site officiel d’Anker SOLIX ainsi que sur Amazon au prix standard de 1299 euros. Cependant, une offre promotionnelle « early bird » sera active du 20 janvier au 23 février 2025, permettant aux acheteurs intéressés d’acquérir cet appareil dès 999 euros ! Cette promotion inclut également un compteur Anker SOLIX Smart offert pour chaque commande passée durant cette période spéciale.
le Solarbank 2 AC représente une avancée significative dans le domaine du stockage énergétique domestique grâce à ses caractéristiques techniques avancées et son engagement envers la durabilité environnementale.
Business
Une formidable nouvelle pour les conducteurs de voitures électriques !
Excellente nouvelle pour les conducteurs de véhicules électriques ! La recharge gratuite sur le lieu de travail sera exonérée d’impôts jusqu’en 2025. Annoncée par le ministère de l’Économie, cette mesure incitative, en place depuis 2020, s’inscrit dans une dynamique de croissance impressionnante avec une progression annuelle moyenne de 35%. Les entreprises peuvent ainsi offrir des bornes de recharge sans impact fiscal, stimulant la transition écologique. Reste à savoir si cela suffira à convaincre les entreprises hésitantes et à propulser l’électrification des flottes professionnelles vers un avenir durable.

Technologie
Recharge Électrique au Bureau : Une Exonération Fiscale Renouvelée
Les détenteurs de véhicules électriques et leurs employeurs peuvent se réjouir : la possibilité d’effectuer des recharges gratuites sur le lieu de travail sera exonérée d’impôts jusqu’en 2025. Cette décision, annoncée par le ministère des Finances, prolonge une initiative lancée en 2020 pour encourager l’adoption des véhicules électriques dans les entreprises.
Un Secteur en Croissance Dynamique
Cette prolongation intervient à un moment clé, alors que le marché des voitures électriques continue d’afficher une croissance remarquable. Entre 2020 et 2022, la progression annuelle moyenne a atteint 35%. En 2023, les particuliers représentent désormais 84% des acquisitions de véhicules électriques, contre seulement 68% en 2018.
Concrètement,cette mesure permet aux sociétés d’installer gratuitement des bornes de recharge pour leurs employés sans impact fiscal. Les frais liés à l’électricité pour ces recharges ne seront pas pris en compte dans le calcul des avantages en nature. De plus, un abattement de 50% sur ces avantages est maintenu avec un plafond révisé à environ 2000 euros pour l’année prochaine.
Accélération Vers une Mobilité Électrique
Cette initiative fait partie d’une stratégie globale visant à promouvoir l’électrification du parc automobile français. Cependant, les grandes entreprises rencontrent encore des difficultés pour atteindre leurs objectifs ; seulement 8% des nouveaux véhicules immatriculés par ces entités étaient électriques en 2023. Ces incitations fiscales pourraient néanmoins inciter davantage d’employeurs à franchir le pas.Cependant, plusieurs défis demeurent concernant les infrastructures nécessaires au chargement ainsi que sur l’autonomie des véhicules et les perceptions parmi les employés. Par ailleurs, la réduction progressive du bonus écologique pour les utilitaires et sa diminution pour les particuliers pourraient freiner cet élan vers une adoption plus large.
Avenir Prometteur Pour La Mobilité Électrique
Malgré ces obstacles potentiels, il existe un optimisme quant au futur de la mobilité électrique dans le milieu professionnel. Les avancées technologiques continues ainsi qu’un engagement croissant envers la durabilité devraient continuer à favoriser cette tendance vers une adoption accrue des véhicules écologiques.
En maintenant ces mesures fiscales avantageuses jusqu’en 2025 et au-delà, le gouvernement délivre un message fort soutenant la transition écologique dans le secteur du transport. Reste maintenant à voir si cela suffira réellement à convaincre certaines entreprises hésitantes et si cela permettra d’accélérer significativement l’électrification de leurs flottes professionnelles dans un avenir proche.
Divertissement
« À la rencontre d’un Hugo : une aventure inattendue »
Le prénom, un véritable reflet de notre identité, peut être à la fois lourd à porter et source de fierté. Dans cette chronique fascinante, le réalisateur Hugo David nous plonge dans son expérience avec un prénom très répandu. Né en 2000, il se retrouve entouré d’autres Hugo, ce qui l’amène à adopter un alias : Hugo D.. Comment ce choix a-t-il influencé son parcours ? Explorez les nuances et les histoires derrière nos prénoms et découvrez comment ils façonnent nos vies dès l’enfance jusqu’à l’âge adulte !

Les Prénoms : Un Voyage au Cœur de l’Identité
Le Rôle Crucial des Prénoms dans nos Existences
Chaque personne possède un prénom, qu’il soit courant ou singulier, et ce dernier peut engendrer à la fois fierté et embarras. Cet article explore la signification profonde et l’influence des prénoms sur notre vie quotidienne. Le réalisateur Hugo David partage son vécu avec un prénom qui a connu une forte popularité durant sa jeunesse.
une Naissance Sous le Signe de la Célébrité
Hugo David est né en 2000 à Tours, une époque où le prénom Hugo était en plein essor. Ses parents, Caroline et Rodolphe, avaient envisagé d’autres choix comme Enzo, également très en vogue à cette période. « Je pense que mes parents ont opté pour un prénom parmi les plus répandus en France plutôt qu’en hommage à Victor Hugo », confie-t-il.
Une Enfance Entourée d’Autres « Hugo »
Dès son plus jeune âge, Hugo se retrouve entouré d’autres enfants portant le même nom. Selon les statistiques de l’Insee,7 694 garçons ont été prénommés Hugo en 2000,faisant de ce prénom le quatrième plus populaire cette année-là. À l’école primaire,il côtoie plusieurs camarades appelés Thibault et autres prénoms similaires. Pour éviter toute confusion lors des appels en classe, les enseignants ajoutent souvent la première lettre du nom de famille après le prénom : ainsi devient-il rapidement « Hugo D. », un surnom auquel il s’habitue sans arduousé.
Pensées sur l’Identité Associée au Prénom
Le choix d’un prénom peut avoir un impact significatif sur notre identité personnelle tout au long de notre existence. Que ce soit pour se distinguer ou pour s’intégrer dans un groupe social spécifique, chaque individu développe une relation particulière avec son propre nom.
les prénoms ne sont pas simplement des désignations ; ils portent avec eux des récits et influencent nos interactions sociales depuis notre enfance jusqu’à l’âge adulte.
-

 Business1 an ago
Business1 an agoComment lutter efficacement contre le financement du terrorisme au Nigeria : le point de vue du directeur de la NFIU
-
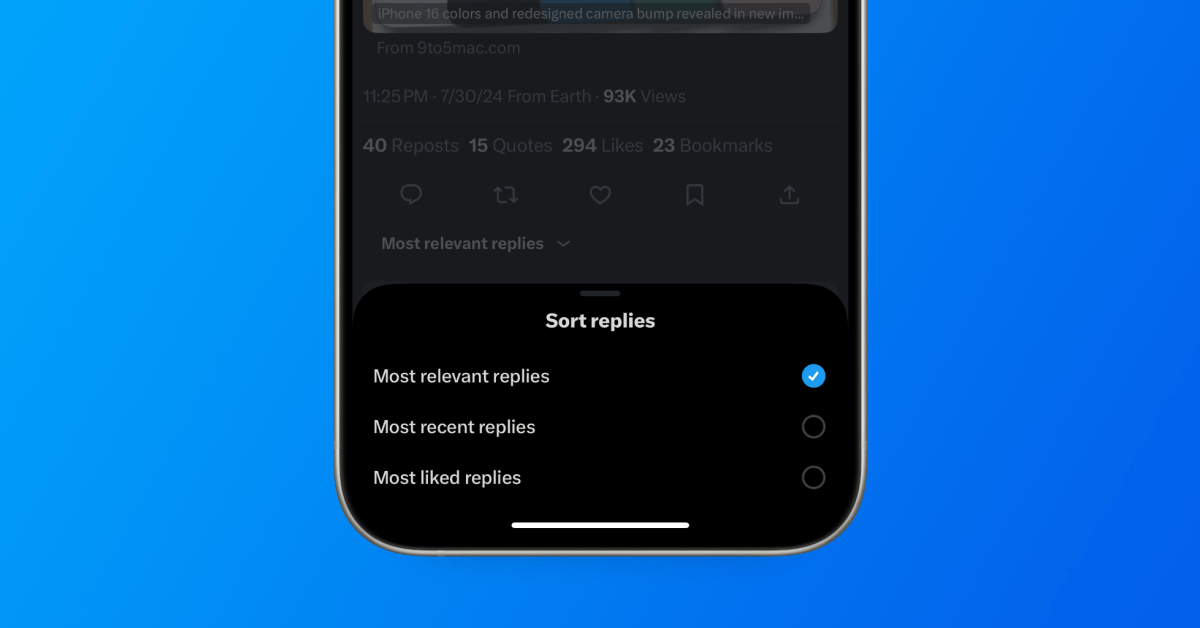
 Général2 ans ago
Général2 ans agoX (anciennement Twitter) permet enfin de trier les réponses sur iPhone !
-

 Technologie1 an ago
Technologie1 an agoTikTok revient en force aux États-Unis, mais pas sur l’App Store !
-

 Général1 an ago
Général1 an agoAnker SOLIX dévoile la Solarbank 2 AC : la nouvelle ère du stockage d’énergie ultra-compatible !
-

 Général1 an ago
Général1 an agoLa Gazelle de Val (405) : La Star Incontournable du Quinté d’Aujourd’hui !
-

 Sport1 an ago
Sport1 an agoSaisissez les opportunités en or ce lundi 20 janvier 2025 !
-

 Business1 an ago
Business1 an agoUne formidable nouvelle pour les conducteurs de voitures électriques !
-

 Science et nature1 an ago
Science et nature1 an agoLes meilleures offres du MacBook Pro ce mois-ci !







