Science et nature
Chai-1 Surpasse AlphaFold 3 : Une Révolution dans la Prédiction des Protéines !
Nous sommes ravis de vous présenter Chai-1, un modèle fondamental multi-modal révolutionnaire pour la prédiction de structures moléculaires. Avec des performances à la pointe de la technologie, Chai-1 permet une prédiction unifiée des protéines, petites molécules, ADN, ARN et bien plus encore. Accessible gratuitement via une interface web, il est également adapté aux applications commerciales telles que la découverte de médicaments. Rejoignez-nous dans cette aventure scientifique et découvrez comment Chai-1 peut transformer vos recherches en biologie !
Technologie
Nous sommes ravis de présenter Chai-1, un nouveau modèle fondamental multi-modal dédié à la prédiction de structures moléculaires, qui se distingue par ses performances de pointe dans divers domaines liés à la découverte de médicaments. Chai-1 permet une prédiction unifiée des protéines, petites molécules, ADN, ARN, modifications covalentes, et bien plus encore.
Le modèle est accessible gratuitement via une interface web, y compris pour des applications commerciales telles que la découverte de médicaments. Nous mettons également à disposition les poids du modèle et le code d’inférence sous forme de bibliothèque logicielle pour un usage non commercial.
Un modèle de pointe pour les interactions biomoléculaires
Nous avons évalué Chai-1 sur un large éventail de benchmarks et avons constaté que le modèle atteint un taux de réussite de 77 % sur le benchmark PoseBusters (contre 76 % pour AlphaFold3), ainsi qu’un Cα LDDT de 0,849 sur l’ensemble de prédiction de structures de monomères de CASP15 (contre 0,801 pour ESM3-98B).
Contrairement à de nombreux outils de prédiction de structures existants qui nécessitent des alignements de séquences multiples (MSA), Chai-1 peut également fonctionner en mode séquence unique sans MSA tout en préservant la majeure partie de ses performances. Le modèle est capable de plier des multimères avec une précision de 69,8 %, surpassant le modèle AlphaFold-Multimer basé sur MSA qui atteint 67,7 %, mesuré par le taux de prédiction acceptable DockQ. Chai-1 est le premier modèle capable de prédire des structures multimères en utilisant uniquement des séquences uniques (sans recherche MSA) avec une qualité équivalente à celle d’AlphaFold-Multimer.
Pour plus de détails et une analyse approfondie du modèle, consultez notre rapport technique.
Un modèle fondamental nativement multi-modal
En plus de ses capacités de modélisation avancées directement à partir des séquences, Chai-1 peut être alimenté avec de nouvelles données, par exemple, des contraintes dérivées de travaux en laboratoire, ce qui améliore les performances de plusieurs points de pourcentage. Nous explorons plusieurs de ces capacités dans notre rapport technique, telles que le conditionnement des épitopes – en utilisant même quelques contacts ou résidus de poches (potentiellement dérivés d’expériences en laboratoire), ce qui double la précision de prédiction des structures anticorps-antigènes, rendant l’ingénierie des anticorps plus réalisable grâce à l’IA.
Mise à disposition du modèle pour tous
Nous mettons Chai-1 à disposition via une interface web gratuitement, y compris pour des applications commerciales telles que la découverte de médicaments. Le code de Chai-1 est également disponible pour un usage non commercial sous forme de bibliothèque logicielle. Nous croyons fermement qu’en collaborant avec les communautés de recherche et industrielles, l’ensemble de l’écosystème en bénéficie.
Essayez Chai-1 par vous-même en visitant lab.chaidiscovery.com, ou exécutez-le depuis notre dépôt GitHub à github.com/chaidiscovery/chai-lab.
Quelles sont les prochaines étapes ?
L’équipe provient de recherches pionnières et d’entreprises d’IA appliquée telles qu’OpenAI, Meta FAIR, Stripe et Google X. Ensemble, nous avons joué des rôles clés dans l’avancement de la recherche en IA pour la biologie. La majorité de l’équipe a été responsable de l’IA dans des entreprises de découverte de médicaments de premier plan et a contribué à faire progresser plus d’une douzaine de programmes de médicaments.
Chai-1 est le fruit de quelques mois de travail intense, et nous ne sommes qu’au début de notre parcours. Notre mission plus large chez Chai Discovery est de transformer la biologie d’une science en ingénierie. Dans cette optique, nous développerons d’autres modèles fondamentaux d’IA capables de prédire et de reprogrammer les interactions entre les molécules biochimiques, les éléments constitutifs fondamentaux de la vie. Nous aurons bientôt plus d’informations à partager à ce sujet.
Nous exprimons notre gratitude envers nos partenaires Dimension, Thrive Capital, OpenAI, Conviction, Neo, Lachy Groom et Amplify Partners, ainsi qu’Anna et Greg Brockman, Blake Byers, Fred Ehrsam, Julia et Kevin Hartz, Will Gaybrick, David Frankel, R. Martin Chavez, et bien d’autres.
Général
Une éruption mystérieuse qui a changé le climat et rendu le soleil bleu : découverte d’une île reculée du Pacifique
Une découverte fascinante a été faite par des chercheurs concernant une éruption volcanique survenue en 1831, qui a modifié le climat et rendu le soleil bleu. En étudiant les cendres dans les carottes de glace polaires, ils ont identifié le Zavaritskii, un volcan isolé des îles Kouriles, comme responsable de cet événement mystérieux. Cette éruption a entraîné un refroidissement climatique de 1 °C, provoquant des famines en Inde et au Japon. William Hutchison, volcanologue à l’Université de St Andrews, souligne que cette découverte révèle encore combien nous ignorons sur l’activité volcanique dans cette région reculée.

Découverte des Origines d’une Éruption Volcanique Historique
Des chercheurs ont récemment élucidé les origines d’une éruption volcanique survenue il y a 200 ans, qui a libéré une quantité massive de soufre dans l’atmosphère, entraînant un changement climatique notable et donnant au soleil une teinte bleue.
Un refroidissement Climatique en 1831
En 1831, la température moyenne dans l’hémisphère nord a chuté d’environ 1,8 degré Fahrenheit (soit environ 1 degré Celsius), coïncidant avec des rapports faisant état de conditions météorologiques sombres et de variations colourées du soleil. Bien que les scientifiques aient établi qu’une éruption volcanique majeure était à l’origine de ce phénomène étrange, le volcan responsable demeurait inconnu jusqu’à présent.
Pistes Découvertes dans les Cœurs Glaciaires
Afin d’identifier la source de cette éruption, les chercheurs ont analysé des cendres retrouvées dans des carottes glaciaires polaires. Leur étude a révélé que l’éruption provenait du volcan Zavaritskii situé sur l’île isolée de Simushir,qui fait partie des îles Kouriles contestées entre la Russie et le Japon. Pendant la guerre froide, l’Union soviétique avait utilisé un cratère volcanique inondé sur Simushir comme base secrète pour sous-marins nucléaires.
Les résultats publiés le 30 décembre 2024 dans la revue PNAS, soulignent combien il reste encore à découvrir concernant l’activité volcanique sur ces îles.
L’impact Météorologique Documenté
« Bien que 1831 soit une période relativement récente, nous n’avions aucune idée que ce volcan était à l’origine [de cette éruption dramatique] », a déclaré William Hutchison, auteur principal et volcanologue à l’Université St Andrews. « C’était complètement hors radar. »
L’éruption de 1831 fait partie d’une série d’éruptions volcaniques au XIXe siècle liées à la phase finale du Petit Âge Glaciaire, qui s’étendait approximativement entre 1800 et 1850. Ce phénomène n’était pas véritablement une période glaciaire — celle-ci ayant pris fin il y a environ dix mille ans — mais représentait néanmoins le moment le plus froid enregistré au cours des cinq derniers siècles.
Météo Dévastatrice en Europe
D’après Hutchison,il n’existe pas de témoignages écrits directs relatant cette éruption en raison du caractère éloigné et peu habité des îles Kouriles souvent enveloppées par un épais brouillard. Cependant, son impact est bien documenté : le compositeur allemand Felix Mendelssohn évoquait un été « désolé » en traversant les Alpes où les températures étaient « aussi froides qu’en hiver ». De nombreux récits rapportent également que le soleil prenait des teintes bleues ou violettes dues aux particules aérosols émises par le panache du volcan. Un phénomène similaire avait été observé après l’éruption du Krakatoa en 1883.

Dérives Mortelles Potentielles
L’éventuelle conséquence mortelle liée à cette éruption pourrait avoir été significative : une baisse globale des températures aurait précédé plusieurs famines majeures en Inde et au Japon durant les années 1830. Hutchison souligne : « Nous savons qu’avec de grandes éruptions volcaniques comme celle-ci, lorsque vous avez un refroidissement climatique cela entraîne aussi des modifications dans les précipitations ainsi que dans les rendements agricoles. » Cela peut engendrer une pénurie alimentaire pour la population.
Mise en Évidence Scientifique Cruciale
Afin d’identifier précisément la source de cet événement cataclysmique passé, Hutchison et son équipe ont examiné les résidus cendreux présents dans diverses carottes glaciaires datant du XIXe siècle prélevées au Groenland. La composition chimique correspondante aux cendres trouvées indiquait clairement leur origine commune avec celles provenant notamment du Japon voisin; cependant ils ont rapidement exclu ce pays comme site potentiel étant donné sa densité démographique élevée ainsi qu’un historique bien documenté concernant ses propres activités volcaniques.
Cela a conduit leurs recherches vers les îles Kouriles.
Sensation ‘Eureka’
L’équipe scientifique découvrit alors que la composition chimique présente aux dépôts cendreux issus du volcano Zavaritskii correspond parfaitement avec celle retrouvée dans ces carottes glaciaires; Hutchison qualifie cela comme étant un moment ‘Eureka’, semblable à celui où on trouve une empreinte digitale lors d’une enquête criminelle.
« C’était vraiment une journée fantastique, » se remémore-t-il avec enthousiasme; « l’une des meilleures journées passées au laboratoire. »
Alerte Manquante Sur Les Volcans Actuels
Tandis qu’ils avaient résolu ce mystère historique lié à l’éruption de 1831 , Hutchison note cependant qu’il n’existe toujours aucun système instrumentalisé surveillant activement toute activité potentielle autour des volcans situés aux îles Kouriles — situation valable pour bon nombre autres volcans autour globe.
« Si cette même éruption devait se produire aujourd’hui, » conclut-il prudemment ; « je ne pense pas que nous serions beaucoup mieux préparés comparativement à notre situation durant année 1831. » Cela illustre combien il sera difficile prédire quand ou où pourrait surgir prochainement un autre événement majeur susceptible modifier notre climat. »
Science et nature
La « reine des loups » de Yellowstone tuée par une meute rivale après 11 ans de vie et 10 portées de louveteaux
Le parc national de Yellowstone a perdu l’une de ses habitantes les plus emblématiques : Wolf 907F, surnommée la « Reine des Loups ». À 11 ans, cette louve au regard unique avait déjà donné naissance à un incroyable nombre de 10 portées. Sa longévité exceptionnelle et son rôle de matriarche en faisaient une figure légendaire. Malheureusement, après une confrontation avec un groupe rival le jour de Noël, sa vie s’est éteinte. Malgré les défis qu’elle a affrontés, Wolf 907F a su mener sa meute avec force et détermination jusqu’à la fin.

Le parc national de Yellowstone a perdu l’un de ses habitants les plus emblématiques et résilients : une louve d’un œil, âgée de 11 ans, qui avait réalisé un exploit en donnant naissance à 10 portées au cours de sa longue vie.
Connue sous le nom affectueux de « Reine des Loups » pour son rôle prépondérant au sein du groupe Junction Butte, la louve 907F est décédée le jour de Noël (25 décembre 2024) suite à une confrontation avec un autre groupe rival.
La louve 907F a vécu plus du double de l’espérance de vie habituelle des loups gris dans le parc, qui est généralement comprise entre quatre et cinq ans. Sa longévité et son statut de matriarche en ont fait une figure légendaire dans l’écosystème du parc ainsi qu’auprès des passionnés d’animaux sauvages.
les événements ayant conduit à sa mort ont débuté le 22 décembre, lorsque la louve 907F et ses petits se nourrissaient d’une carcasse de bison sur la rive nord du fleuve Yellowstone, d’après Cowboy State Daily.Des membres du groupe rival Rescue Creek — habituellement confinés sur la rive sud — ont traversé le fleuve sans prévenir et se sont engagés dans une violente altercation avec le groupe dirigé par 907F.
Bien que la louve ait survécu à cette première rencontre, ses blessures se sont révélées fatales. Son collier radio, surveillé par les biologistes du parc Yellowstone, a signalé son immobilité le 26 décembre, indiquant qu’elle était probablement décédée la veille.
Une des plus anciennes louves de Yellowstone

Avec ses onze années passées dans ce milieu sauvage depuis leur réintroduction en 1995 au parc national yellowstone,d’après Smithsonian Magazine, elle faisait partie des six rares spécimens ayant atteint cet âge avancé.
Louve courageuse face aux défis rencontrés tout au long de sa vie. Les scientifiques pensent qu’elle aurait perdu son œil gauche vers l’âge four; cependant, les raisons exactes demeurent floues. de plus, elle avait survécu à un épisode sévère dû à la gale — maladie cutanée contagieuse causée par des acariens microscopiques pouvant être mortels.

Même face aux adversités rencontrées tout au long des années passées en tant que leader efficace pour son groupe familial , elle a réussi à donner naissance encore une fois lors du printemps dernier (2024), surprenant ainsi les chercheurs par sa vitalité malgré son âge avancé.
Ayant mis bas chaque année durant toute une décennie avant sa mort dès qu’elle atteignit maturité sexuelle , Rabe affirmait auprès du Cowboy State Daily que cela faisait d’elle « la femelle ayant connu le succès reproductif sans précédent dans toute l’histoire connue ».
Science et nature
Les meilleures offres du MacBook Pro ce mois-ci !
Apple a récemment dévoilé ses nouveaux modèles de MacBook Pro de 14 et 16 pouces, équipés des puissants processeurs M4, M4 Pro et M4 Max. Ces modèles améliorés, lancés en novembre 2024, offrent le double de RAM par rapport à leurs prédécesseurs. Si vous envisagez d’acheter un MacBook Pro, sachez que les anciens modèles M3 sont désormais en liquidation avec des remises intéressantes. Cependant, attention à la différence de RAM ! Découvrez nos conseils pour dénicher les meilleures offres et faire le choix parfait pour vos besoins.

Les Nouveaux Modèles de MacBook Pro : Ce Qu’il Faut Savoir
En octobre 2024, Apple a dévoilé ses derniers modèles de MacBook Pro de 14 et 16 pouces, équipés des puces M4, M4 Pro et M4 Max. Ces nouvelles versions arrivent un an après le lancement des variantes M3, qui avaient déjà fait sensation. Les nouveaux modèles offrent une double capacité de RAM par rapport à leurs prédécesseurs.
Les ventes ont débuté le vendredi 8 novembre et les appareils sont désormais disponibles à l’achat.
Acheter un MacBook Pro : Faut-il opter pour les anciens modèles ?
Si vous envisagez d’acheter un MacBook Pro, vous pourriez être tenté par les modèles de la série M3 qui pourraient bénéficier de réductions alors que les revendeurs écoulent leur stock.Cependant, gardez à l’esprit que ces anciens modèles disposent d’une moitié moins de RAM que les nouveaux. Apple facture environ 200 $/200 £ pour une mise à niveau de la RAM au moment de l’achat ; il est donc essentiel que toute réduction sur un ancien modèle prenne cela en compte. De plus, si vous êtes en dehors des États-Unis, notez que le prix des nouveaux modèles peut être inférieur à celui des anciens.
avec une combinaison d’augmentations et diminutions tarifaires dans toute la gamme par rapport aux générations précédentes, il peut s’avérer difficile d’identifier une bonne affaire. Heureusement, notre équipe d’experts apple scrute Internet tout au long de l’année pour dénicher les meilleures offres sur le MacBook pro ainsi que sur le MacBook Air qui présente également plusieurs similitudes avec son homologue pro.
Comparaison des Prix Actuels
Vous trouverez ci-dessous des tableaux comparatifs affichant les meilleurs prix actuels pour chaque modèle standard du MacBook Pro (y compris ceux mis en vente depuis le 8 novembre). Des offres sont également disponibles pour certains modèles désormais discontinués tant qu’ils restent en stock chez divers détaillants.
Aperçu des Meilleures Offres Actuelles
- M4 – MacBook Pro 14 pouces :
- Amazone : modèle avec 512 Go/16 Go RAM – $1,459 (140 $ off)
- Amazone : Modèle avec 1 To/24 Go RAM – $1,819 (180 $ off)
- Amazone : Modèle M4 Pro avec 1 To/24 Go RAM – $2,179 (220 $ off)
- Amazone : Modèle M4 Max avec 16 pouces – À partir de $3,174 (325 $ off)
Détails Sur Les Offres Discontinuées
- M3 – ancien modèle :
- B&H photo: Modèle avec 512 Go/18 Go RAM – Environ $1,599 (-400$)
- Best Buy: Version précédente du modèle pro – Environ $2,199 (-300$)
L’Importance D’un Guide D’Achat Complet
Pour obtenir davantage d’informations et choisir votre ordinateur portable Apple idéalement adapté à vos besoins spécifiques ou professionnels , consultez notre guide complet où nous abordons tous les différents types disponibles ainsi qu’un guide spécifique aux achats concernant toute la gamme Macintosh.
Démarche Pour Acquérir Un Nouveau Produit Apple
Tandis qu’il est possible d’acquérir directement auprès d’Apple ou via divers revendeurs agréés dans le monde entier — beaucoup offrant aussi bien sur produits neufs qu’anciens — il vaut toujours mieux comparer avant achat afin maximiser vos économies potentielles ! Vous pouvez consulter nos recommandations concernant où acheter votre prochain appareil Macintosh ici même !
Détaillants Recommandés Aux États-Unis :
- Boutique officielle Apple
- Boutique Éducation Apple
- Amazone
< li>B&H Photo < li > Best Buy < / li >< li > Walmart < / li >< / ul >
< h r >
< h r >
< p > Si vous recherchez encore plus moyen économe , découvrez si acheter vos produits électroniques depuis un autre pays pourrait réduire considérablement votre budget .< / p >
< h r >
< h r >
< h o u t e s t i n g = "u k retailers" >Détaillants Recommandés Au Royaume-Uni:
< ul >< li > Boutique officielle Apple< / li >< li > Boutique Éducation Apple< / li >< li > Amazone< / li >< < ...
-

 Business1 an ago
Business1 an agoComment lutter efficacement contre le financement du terrorisme au Nigeria : le point de vue du directeur de la NFIU
-
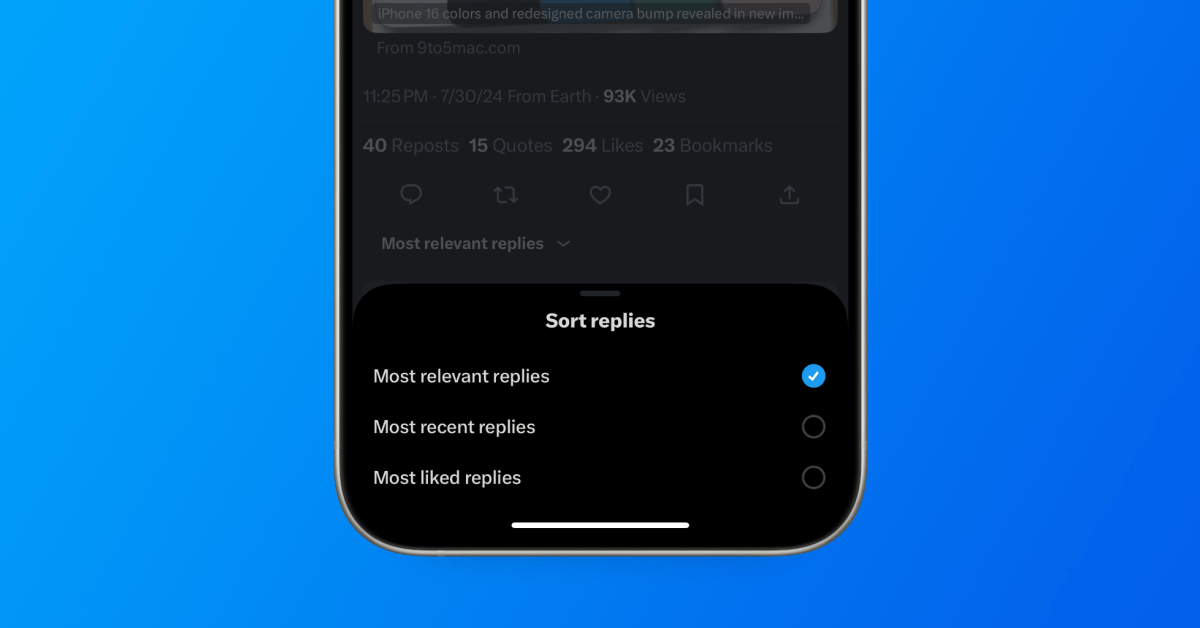
 Général2 ans ago
Général2 ans agoX (anciennement Twitter) permet enfin de trier les réponses sur iPhone !
-

 Technologie1 an ago
Technologie1 an agoTikTok revient en force aux États-Unis, mais pas sur l’App Store !
-

 Général1 an ago
Général1 an agoAnker SOLIX dévoile la Solarbank 2 AC : la nouvelle ère du stockage d’énergie ultra-compatible !
-

 Général1 an ago
Général1 an agoLa Gazelle de Val (405) : La Star Incontournable du Quinté d’Aujourd’hui !
-

 Sport1 an ago
Sport1 an agoSaisissez les opportunités en or ce lundi 20 janvier 2025 !
-

 Business1 an ago
Business1 an agoUne formidable nouvelle pour les conducteurs de voitures électriques !
-

 Science et nature1 an ago
Science et nature1 an agoLes meilleures offres du MacBook Pro ce mois-ci !