Santé et médecine
Des scientifiques révèlent comment les bases de données peuvent révolutionner la sécurité alimentaire !
Des avancées prometteuses pour la sécurité alimentaire ! Les scientifiques affirment qu’une base de données innovante, créée dans le cadre du projet européen MASTER, pourrait révolutionner notre approche de la sécurité alimentaire. La cFMD, qui regroupe des métagénomes alimentaires, permet une analyse rapide et précise des microorganismes présents dans nos aliments. Grâce à des technologies de séquençage ADN, cette ressource inestimable aide à identifier et contrôler les microbes indésirables, tout en soutenant la qualité et la durabilité des produits alimentaires. Une véritable révolution pour l’industrie alimentaire !

Amélioration de la Sécurité Alimentaire grâce à une Base de Données Innovante
Une avancée significative dans la recherche alimentaire
Des chercheurs affirment qu’une base de données développée dans le cadre d’un projet européen pourrait renforcer la sécurité alimentaire. Cette base de données, qui regroupe des métagénomes alimentaires, a été créée dans le cadre du projet MASTER financé par l’UE. Les métagénomes représentent le matériel génétique de tous les microorganismes présents dans un environnement donné.
La base de données cFMD
La base de données, nommée cFMD (curated Food Metagenomic Data), compile des métagénomes issus de divers aliments. Les technologies de séquençage de l’ADN permettront aux chercheurs de s’attaquer à des problématiques telles que le gaspillage alimentaire, la résistance aux antimicrobiens et la sécurité des aliments. Les microorganismes peuvent avoir des effets bénéfiques sur la production alimentaire, comme dans le cas de la fermentation, mais peuvent également causer des problèmes tels que la détérioration des aliments ou des maladies d’origine alimentaire.
Méthodes d’analyse traditionnelles
L’analyse de ces microorganismes reposait auparavant sur des méthodes traditionnelles nécessitant la culture des microorganismes sur des milieux gélosés dans des plaques de Petri, avec des milieux spécifiques pour chaque type de microorganisme.
Avantages pour la sécurité alimentaire
Les scientifiques estiment que l’utilisation de méthodes de séquençage de l’ADN à haut débit pourrait révolutionner les tests alimentaires. Cette approche permet un test rapide et simultané de tous les microorganismes, y compris ceux qui sont difficiles à cultiver. Des milliers d’échantillons prélevés dans des installations de transformation alimentaire en Europe ont été soumis à des tests ADN. Grâce à cette base de données, les résultats des analyses basées sur le séquençage de l’ADN peuvent être traités rapidement et avec précision pour identifier et contrôler les microorganismes indésirables.
Détails du projet MASTER
Le projet MASTER, qui a débuté en janvier 2019, regroupe 30 partenaires et s’étendra sur quatre ans. Il utilise des technologies de séquençage pour cartographier les microbiomes dans divers environnements alimentaires et non alimentaires. Le professeur Paul Cotter, coordinateur du projet et responsable des biosciences alimentaires à Teagasc (l’Autorité irlandaise de développement de l’agriculture et de l’alimentation), a indiqué que les données couvrent 15 catégories alimentaires provenant de 52 pays.
Une ressource précieuse pour les scientifiques
Le cFMD contient des données sur 3 600 espèces microbiennes, dont 290 sont nouvelles. Cette base de données est accessible gratuitement et peut être largement utilisée pour des études sur les microbiomes et des applications dans l’industrie alimentaire. Par exemple, elle peut aider à étudier le déplacement des microbes le long de la chaîne alimentaire, à analyser la propagation des gènes de résistance aux antimicrobiens, à détecter des microbes indésirables et à examiner la transmission des microbes à l’homme. La disponibilité du cFMD représente une avancée majeure vers un avenir où le séquençage métagénomique pourrait remplacer la microbiologie classique comme outil de suivi des microbes dans la chaîne alimentaire.
Recherches connexes
Une étude récente publiée dans la revue Cell a été menée par des équipes de l’Université de Trento et de l’Université de Naples Federico II en Italie, ainsi que par Teagasc d’Irlande, le Conseil national de la recherche espagnol et l’Université de León en Espagne, MATIS d’Islande et FFoQSI en Autriche, avec d’autres contributeurs.
Le professeur Danilo Ercolini du Département des sciences agricoles de l’Université de Naples Federico II a déclaré : « La disponibilité d’un ensemble de données aussi vaste, incluant des métagénomes alimentaires et des génomes de microbes alimentaires, sera une ressource essentielle pour de nombreux scientifiques de l’alimentation afin d’explorer davantage la présence et le rôle des microbes dans les aliments et les installations de transformation alimentaire, dans le but ultime d’améliorer la qualité, la sécurité et la durabilité des aliments. »
Observations sur les espèces microbiennes
Bien que la plupart des espèces connues pour leur transmission alimentaire aient été rarement détectées dans les aliments échantillonnés, Listeria monocytogenes a été identifiée une fois, et Clostridium perfringens a été trouvé à trois reprises. Les chercheurs ont également découvert des centaines d’espèces non caractérisées dans le microbiome alimentaire.
« L’isolement ciblé et la caractérisation fonctionnelle de ces microbes alimentaires que nous avons détectés uniquement par métagénomique devraient être la prochaine étape pour exploiter davantage leur rôle dans le traitement, la qualité et la sécurité des aliments. Nos résultats soutiennent le typage moléculaire, ce qui signifie développer des certifications d’authenticité et d’origine des aliments basées sur la spécificité microbienne », ont-ils ajouté.
Général
Le Rotary investit 2 millions de dollars pour la santé maternelle : le président de la Fondation en visite au Nigeria pour explorer de nouvelles opportunités
Erreur 403 : Accès refusé !
Vous êtes face à un mur numérique. Impossible d’accéder à ce document, et la frustration monte. Que faire dans ces moments-là ? Ne paniquez pas ! Voici quelques solutions simples :
- Cliquez sur « Recharger la page »
- Passez à la page précédente
- Retournez à l’accueil
N’oubliez pas, chaque obstacle est une opportunité d’explorer de nouvelles voies !

Erreur de Serveur : Accès Refusé
Vous n’avez pas l’autorisation d’accéder à ce document.
Voici quelques actions possibles :
Rafraîchir la page
Retourner à la page précédente
Accéder à la page d’accueil
Santé et médecine
Les refuges d’Ottawa : Dîners festifs et cadeaux pour les résidents pendant les fêtes !
Le jour de Noël, les Pasteurs de la Bonne Espérance ont accueilli des centaines de personnes pour un repas festif chaleureux. Azery Sharrons, un homme de 70 ans vivant au refuge, a exprimé sa gratitude pour ce repas qui lui rappelle des souvenirs d’enfance. Husa Delice, un bénévole dévoué depuis 2022, a souligné l’importance de cette communauté où les sourires et la chaleur humaine règnent. Ce Noël n’était pas seulement une fête; c’était une occasion d’unir ceux qui en ont besoin et d’apporter un peu de réconfort dans leurs vies difficiles.

Célébrations de Noël au Refuge des Bons Samaritains
un repas chaleureux pour les plus démunis
Le jour de Noël, le Refuge des Bons samaritains a accueilli un grand nombre de personnes pour un repas fait maison. Les organisateurs s’attendaient à servir plusieurs centaines de convives tout au long de la journée.
L’engagement des bénévoles
Husa Delice, bénévole depuis 2022, a partagé son expérience en disant que cet endroit était « merveilleux » et lui avait permis de rencontrer des gens formidables. Il a remarqué que les membres de la communauté étaient »heureux » et « sourire aux lèvres ».
Une atmosphère conviviale malgré le froid
Delice a souligné l’importance d’offrir chaleur et réconfort aux personnes dans le besoin pendant cette période froide. Azery Sharrons, un homme âgé de 70 ans vivant dans une zone d’accueil du refuge, a exprimé sa gratitude pour le repas copieux qui rappelait les traditions festives.
Activités post-Noël au refuge
Le lendemain du jour de Noël, le refuge prévoit d’accueillir un groupe de chanteurs qui viendront égayer l’après-midi avec des chants traditionnels. Des dons tels que vêtements chauds et produits d’hygiène seront également distribués aux visiteurs.
Besoins matériels essentiels
Les dons comme tasses, assiettes et couverts sont cruciaux pour la cuisine communautaire. De plus, les couvertures et oreillers sont très appréciés par ceux qui séjournent au refuge.
Élargissement des festivités
En parallèle à ces activités sur leur site principal rue Murray, l’équipe du Refuge des Bons Samaritains s’est rendue dans cinq résidences offrant un logement soutenu afin d’y servir également le dîner traditionnel ainsi que distribuer des cadeaux.
Préparatifs minutieux en cuisine
Peter Gareau, responsable du service alimentaire au refuge, a expliqué qu’une préparation intensive est nécessaire avant Noël.Bien qu’il ait initialement craint ne pas avoir assez de dindes à cause du nombre élevé attendu, il a été soulagé par la générosité locale qui lui a permis d’obtenir suffisamment pour nourrir tous les invités durant toute la semaine.
Gareau souligne aussi que ces repas festifs peuvent raviver chez certains clients des souvenirs heureux liés à leur enfance : « Personne ne choisit ce mode de vie », dit-il en espérant que cela puisse inciter certains à participer aux programmes offerts par l’établissement.
Autres initiatives communautaires
D’autres organisations ont également organisé leurs propres célébrations durant cette période festive.Cornerstone Housing for Women par exemple avait prévu une collecte communautaire permettant la création de 321 sacs-cadeaux destinés aux résidents en situation précaire.
Chris O’Gorman mentionne que ces gestes montrent clairement aux bénéficiaires qu’ils sont soutenus par leur communauté. En collaboration avec Restaurant 18, ils ont préparé plusieurs centaines de plats traditionnels pour ceux vivant dans leurs installations.
Dons inattendus
De son côté Peter Tilley, directeur général du Ottawa Mission (Mission d’Ottawa), raconte comment ils ont reçu plus de 200 dons provenant d’un groupe appelé Backpacks for the Homeless (Sacs à dos pour sans-abri). Ces paquets contenaient divers articles allant des chaussettes jusqu’à une carte-cadeau Tim Hortons.
Tilley ajoute qu’ils ont ouvert leur chapelle toute la journée afin que ceux qui se sentent seuls puissent regarder tranquillement quelques films classiques tout en profitant ainsi d’un moment réconfortant pendant cette période difficile.
Santé et médecine
Les étoiles de Downton Abbey : que sont-elles devenues depuis la fin de la série ?
Il y a presque neuf ans, Downton Abbey a tiré sa révérence sur ITV, mais l’héritage de cette série emblématique perdure. Grâce à deux films et un troisième en cours de tournage, les fans peuvent retrouver leurs personnages préférés. Hugh Bonneville, Michelle Dockery et Joanne Froggatt ont continué à briller sur nos écrans avec des rôles captivants. Découvrez ce que ces acteurs talentueux ont accompli depuis la fin de la série et comment ils ont évolué au fil des années. La magie de Downton Abbey n’est pas prête de s’éteindre !
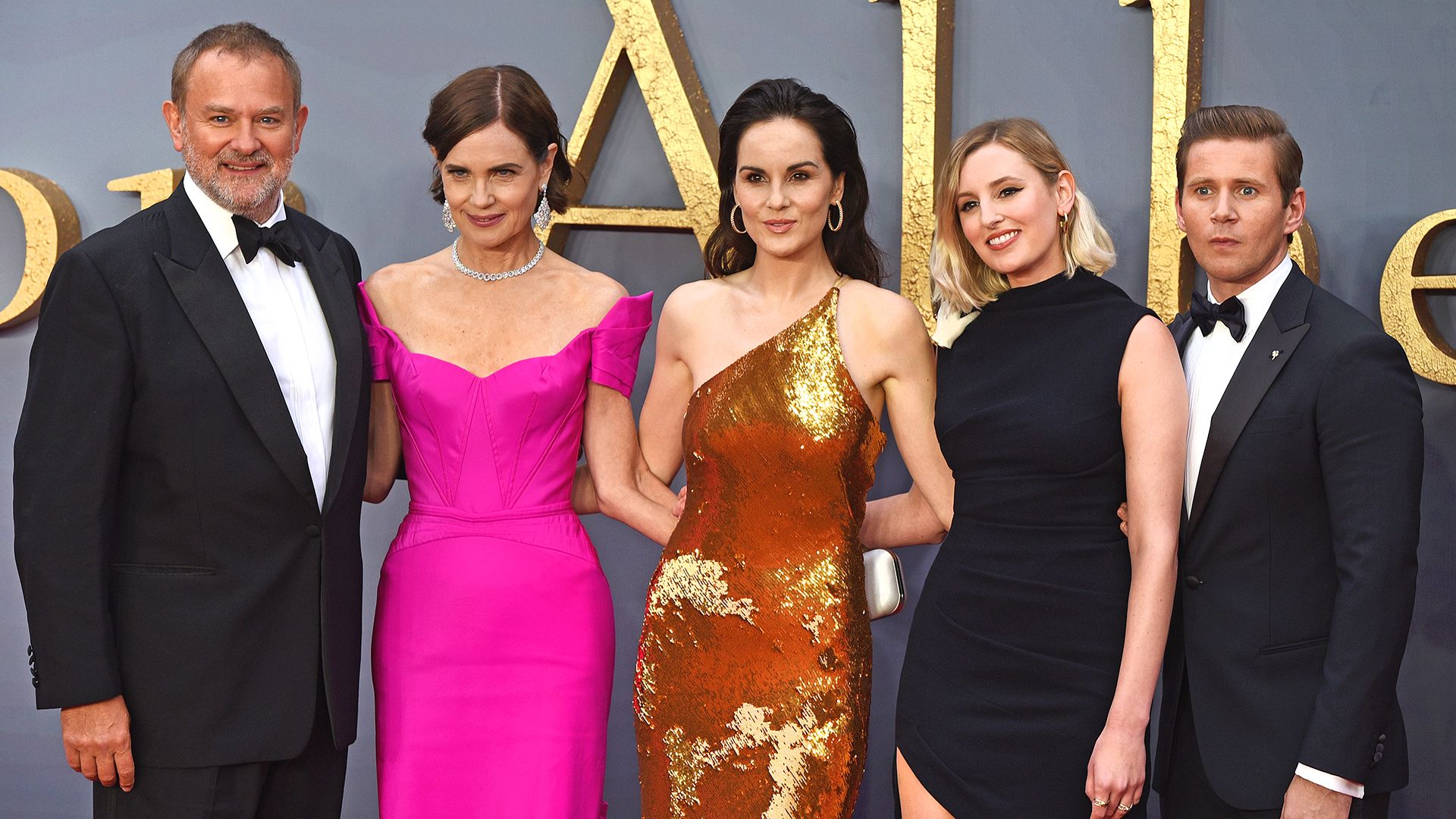
Medecine
Le temps passe vite, cela fait déjà neuf ans que la série Downton Abbey a pris fin sur ITV. Heureusement,cette franchise a donné naissance à deux films,et un troisième a été tourné cet été.
Depuis la conclusion de la série, nous avons eu le plaisir de voir les acteurs reprendre leurs rôles emblématiques, bien que quelques exceptions notables soient à signaler. Bien que nous n’ayons jamais vraiment dit adieu aux personnages, beaucoup de choses ont évolué pour Hugh Bonneville, Michelle Dockery, Joanne Froggatt et d’autres depuis leur passage dans l’émission. Voici un aperçu de ce qu’ils ont réalisé au cours des presque dix dernières années…
### Hugh Bonneville : Un parcours diversifié
Hugh Bonneville est reconnu pour son interprétation du comte Robert Crawley. Après la fin de Downton Abbey, il a continué à briller sur nos écrans dans la franchise Paddington, tout en décrochant des rôles dans des productions telles que I Came By (2022), Bank of Dave (2023) et le prochain film intitulé douglas is Cancelled, prévu pour 2024.
Cet été, Hugh a également participé au tournage du troisième film de la saga Downton Abbey qui n’a pas encore reçu son titre officiel.
### Michelle Dockery : Des rôles audacieux
Michelle Dockery continue d’incarner Lady Mary Crawley avec brio tout en s’attaquant à des rôles plus sombres et complexes. Parmi ses récentes apparitions télévisées figurent les séries godless (2017), Anatomy of a Scandal (2022) et le futur projet intitulé This Town, prévu pour 2024. Au cinéma, elle est apparue dans des films tels que The Gentlemen (2019), ainsi que dans les productions récentes comme Boy kills World (2023) et le très attendu film < em >Flight Risk (2024).
### Joanne Froggatt : Une carrière florissante
Joanne Froggatt est devenue célèbre grâce à son rôle d’Anna Bates dans la série acclamée ainsi que dans ses adaptations cinématographiques. Depuis l’arrêt du show en 2015, elle a obtenu des premiers rôles dans plusieurs séries télévisées telles que < em >Liar (2017-20), < em >Angela Black (2021) et < em >Breathtaking (prévu pour 2024). Récemment confirmée pour participer à une nouvelle série criminelle sans titre réalisée par Guy Ritchie aux côtés de Tom Hardy et Pierce brosnan.
### Jessica Brown findlay : Une étoile montante
Jessica Brown Findlay s’est fait connaître en incarnant Lady Sybil Branson avant de quitter Downton Abbey en 2012. Depuis lors, elle a joué dans plusieurs productions notables comme < em >jamaica Inn (2014), < em >Harlots (2017-19), ainsi qu’< emp >Brave New World (2020). Son dernier projet inclut une apparition prévue dans la série télévisée Playing Nice avec James Norton qui sortira le 5 janvier prochain.
### Laura Carmichael : L’artiste polyvalente
Laura Carmichael s’est illustrée par sa performance remarquable en tant qu’Edith Crawley. elle continue d’apporter sa touche unique au petit écran avec notamment son rôle marquant en tant que Margaret Pole dans < emp>The Spanish princess (2019-20) ou Agatha dans < emp>The Secrets She Keeps .
### Jim Carter : Un acteur prolifique
Jim Carter ne chôme pas ! après l’arrêt de Downton Abbey , il est apparu sur scène notamment avec sa participation remarquée à king Lear (2018) ou encore The Good Liar (2019). Plus récemment il était présent au cinéma avec The Sea Beast (2022) puis Wonka (prévu pour 2023).
### Penelope Wilton : Une carrière impressionnante
Penelope Wilton est célébrée pour son interprétation d’Isobel Gray .Sa filmographie comprend diverses œuvres telles que The Guernsey Literary and Potato Peel Pie Society(2018), After Life(2019-22 ), Operation Mincemeat( prévus cette année )et The Unlikely Pilgrimage of Harold fry( également prévu cette année ).
### Hommage à Dame Maggie Smith
Maggie Smith brillait sous les traits puissants de Violet Crawley , Comtesse douairière de Grantham . En plus d’avoir repris ce rôle iconique lors des films suivants,
Parmi ses nombreux projets récents figurent Sherlock Gnomes(18 ), A Boy Called Christmas(en21 )etThe Miracle Club(en23 ). Malheureusement,maggie nous a quittés le27 septembre dernierà l’âgede89 ans,soulevant une vagued’hommages émouvantsde lamisde Downtown.
-

 Business1 an ago
Business1 an agoComment lutter efficacement contre le financement du terrorisme au Nigeria : le point de vue du directeur de la NFIU
-
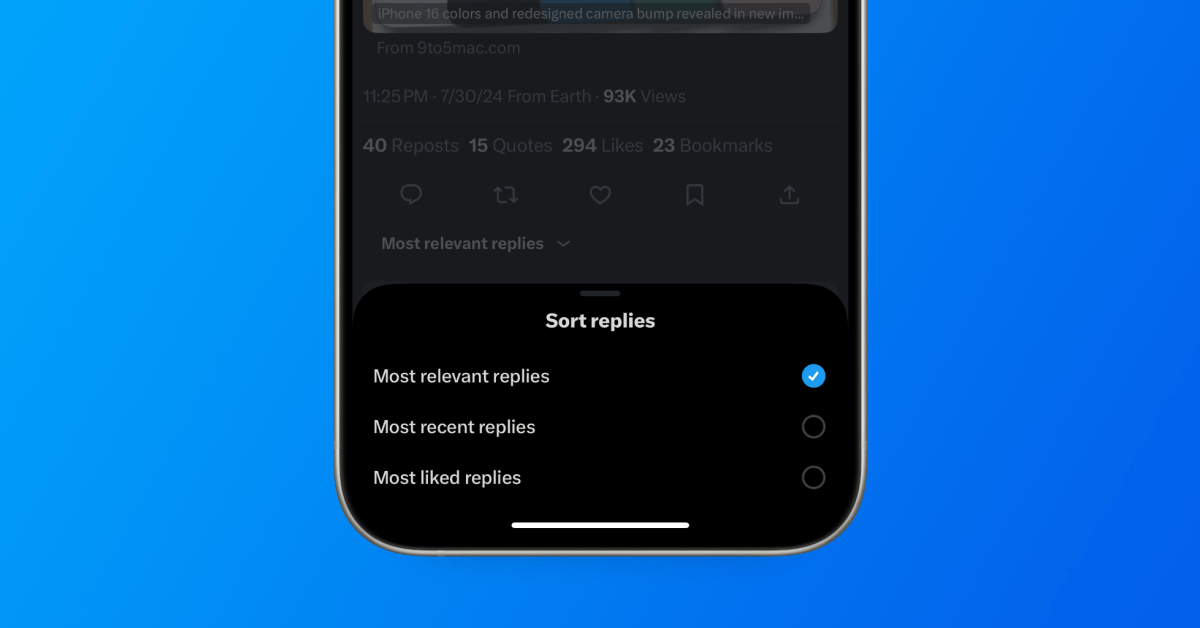
 Général2 ans ago
Général2 ans agoX (anciennement Twitter) permet enfin de trier les réponses sur iPhone !
-

 Technologie1 an ago
Technologie1 an agoTikTok revient en force aux États-Unis, mais pas sur l’App Store !
-

 Général1 an ago
Général1 an agoAnker SOLIX dévoile la Solarbank 2 AC : la nouvelle ère du stockage d’énergie ultra-compatible !
-

 Général1 an ago
Général1 an agoLa Gazelle de Val (405) : La Star Incontournable du Quinté d’Aujourd’hui !
-

 Sport1 an ago
Sport1 an agoSaisissez les opportunités en or ce lundi 20 janvier 2025 !
-

 Business1 an ago
Business1 an agoUne formidable nouvelle pour les conducteurs de voitures électriques !
-

 Science et nature1 an ago
Science et nature1 an agoLes meilleures offres du MacBook Pro ce mois-ci !











